A aquisição eletrônica de dados passa a disponibilizar, em pouco tempo, um grande volume de dados criando outro desafio, o tratamento adequado dos dados para a obtenção de informações úteis. Para a manipulação eficiente de grande volume de dados é necessário o uso da Quimiometria, que pode ser definida como: “uma disciplina química que emprega métodos matemáticos, estatísticos e computacionais para planejar ou selecionar experimentos de forma otimizada e para fornecer o máximo de informação química com a análise dos dados obtidos”.
Um marco importante no surgimento da quimiometria foram os artigos publicados em 1969 por Jurs, Kowalski, Isenhour e Reilly dentre os quais podemos citar: Computerized Learning Machines applied to Chemical Problems, Multicategory pattern classification by least squares, 1969 e Computerized Learning Machines applied to Chemical Problems, Investigation of combined patterns from diverse analytical data using computerized learning machines, 1969.
Mas o termo “Quimiometria” foi usado pela primeira vez em 1971 pelo químico orgânico Svante Wold, no editorial do primeiro exemplar do Journal of Chemometrics. (Quimiometria - Conceitos, Métodos e Aplicações, 2015)
Para que a análise de um conjunto de dados seja bem-sucedida, deve-se seguir um roteiro constituído das seguintes etapas:
Organização, visualização e pré-tratamento dos dados
Análise exploratória dos dados
E dependendo do objetivo final:
Construção de modelos de classificação (qualitativos ou categóricos)
Construção de modelos de regressão (quantitativos)
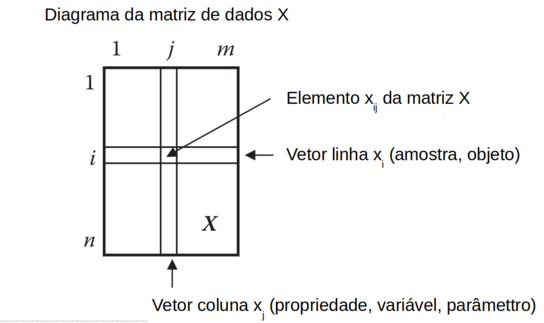
Os dados multivariados são usualmente organizados na forma de tabela (matriz, planilha) formada por “n” linhas e “m” colunas.
Cada linha corresponde a uma “amostra” (objeto), e cada coluna corresponde a um “atributo” (propriedade, variável, parâmetro) analisado (medido) da respectiva amostra.
Nessa matriz, indicada por “X”, cada elemento da matriz é indicado por “xij” onde “i” é a linha, e “j” a coluna. (Figura R.1)
Figura R.1. Diagrama da matriz X, de dados multivariados, com “n” linhas (amostras, objetos) e “m” colunas (propriedades, variáveis, parâmetros, característica)
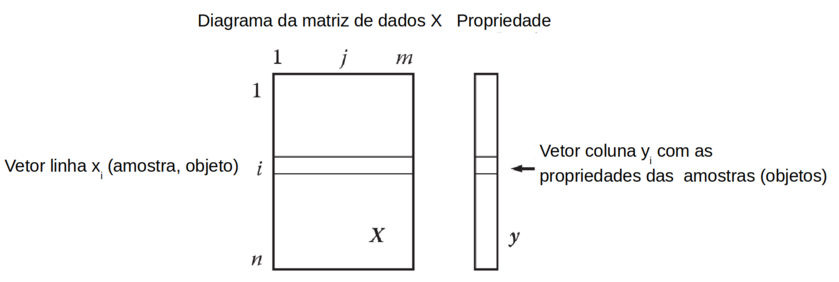
Além da matriz “X” é necessário uma matriz de uma (ou mais) coluna “Y” com as respectivas propriedades (ou rótulos) para cada amostra. (Figura R.2).
Figura R.2. Matriz “X” e a respectiva matriz coluna Y com as respectivas “propriedades” “yi” (resposta) para cada variável “xi”. As propriedades (respostas) podem ser variáveis contínuas ou discretas. (Fonte: Introduction to multivariate statistical analysis in chemometrics, 2009)
Nota
A figura R.2 mostra um exemplo de uma estrutura de dados “bidimensional”. Mas é importante lembrar que existem estruturas de dados com maior “dimensionalidade”.
Imagine que cada elemento da matriz X (xij) não seja um número mas seja um vetor com N elementos. Nesse caso teremos uma estrutura “tridimensional” de dados.
Um exemplo é o conjunto dos espectros de emissão de fluorescência em diferentes comprimentos de onda de excitação para diferentes amostras. Nesse caso cada linha da matriz X seria correspondente a uma amostra, cada coluna da matriz X seria correspondente aos diferentes comprimentos de onda de excitação, e cada elemento xij da matriz X seria um vetor com as intensidades de emissão para diferentes comprimentos de onda.
Uma vez organizados na forma de matriz, é muito importante visualizar os dados por meio de gráficos e verificar como estão distribuídos antes de iniciar qualquer análise.
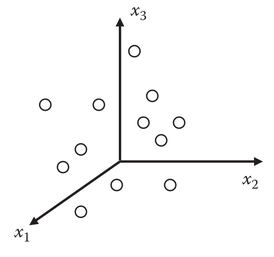
Geometricamente, cada amostra (objeto) pode ser considerado como um “ponto” em um espaço com m dimensões, onde cada dimensão (coordenada) representa uma propriedade (variável, parâmetro, característica).
Por exemplo, a figura R.3 representa um espaço tridimensional (m=3) no qual cada coordenada (eixo) representa uma variável (x1, x2 e x3) e cada ponto no espaço representa uma amostra (objeto).
Figura R.3. Gráfico que mostra cada amostra (objeto) em um espaço 3D onde cada dimensão (x1, x2 e x3) corresponde a uma variável (propriedade, parâmetro, característica)(Fonte: Introduction to multivariate statistical analysis in chemometrics, 2009)
Todo sinal registrado por um instrumento possui um “erro associado”, que pode ser:
Erro Sistemático - No qual todas as medidas são sistematicamente menor ou maior do que o valor verdadeiro.
Erro Aleatório - No qual existem variações imprevisíveis no sinal medido de momento a momento ou de medição a medição.
Esse tipo de erro é comumente chamado de “ruído”.
O Ruído pode ser reduzido, mas não eliminado.
Como o sinal do Ruído pode ser negativo ou positivo, pode ser reduzido adicionando-se medidas repetidas.
Além do ruído aleatório o sinal analítico também pode conter “um erro sistemático” como, por exemplo, o deslocamento “constante” da “linha de base” do sinal (offset) causado pelo instrumento (Ex: perda de sensibilidade do sensor ao longo do tempo, luz espúria em um fotômetro) ou pela própria amostra (Ex: turbidez).
Ou o deslocamento “variável” da “linha de base” do sinal (bias, viés, vício, tendência) (Ex: mudanças na temperatura durante a análise, oscilações elétricas).
O objetivo do pré-tratamento é reduzir as variações indesejáveis nos dados e que podem influenciar os resultados finais.
Mas é importante avaliar o impacto da técnica utilizada no pré-tratamento para não distorcer ou comprometer os resultados da análise.
Há dois tipos de pré-tratamento:
Transformação: aplicado às amostras (linhas da matriz X)
Pré-processamento: aplicado às variáveis (colunas da matriz X)
A "transformação dos dados" (ao longo das linhas da matriz) é aplicada quando os parâmetros analisados para cada amostra do mesmo “tipo” (natureza), por exemplo, um espectro com as intensidades de absorbância em função do comprimentos de onda, um cromatograma com as intensidades do detector em função do tempo.
Enquanto que o "pré-processamento dos dados" (ao longo das colunas da matriz X) é aplicado quando são diferentes tipos de parâmetros para cada amostra como, por exemplo, as concentrações de diferentes íons em amostras de água.
Alguns exemplos de “transformação de dados”:
Adição, subtração, multiplicação ou divisão de espectros (Signal arithmetic)
Alisamento de espectros (Smoothing)
Derivação (diferenciação) para correção da linha de base (Derivação)
As técnicas de pré-processamento são aplicadas às variáveis (parâmetros, propriedades, fatores, características), ou seja, a cada uma das colunas da metriz de dados X.
Os dados originais podem não ter uma “distribuição” adequada dificultando a extração de informações e a interpretação dos mesmos. Por exemplo, quando duas ou mais variáveis (fatores) possuem valores numéricos (em diferentes unidades) ou diferentes variâncias.
Nesses casos é necessário fazer um pré-processamento dos dados originais usando as técnicas de: centralização (centrar na média), escalamento pela variância e/ou autoescalamento (ou padronização ou transformação z).
Dependendo da situação podem ser aplicadas apenas uma ou ambas as técnicas.
A centralização é feita calculando a média de cada variável e subtraindo-se cada variável do respectivo valor médio (Equação R.1).
Com a centralização as variáveis passam a ter média igual a “0”.
No método de escalamento, as variáveis são divididas pelo respectivo desvio padrão (Equação R.2).
E o autoescalamento (ou padronização) é a combinação da “centragem na média” com o “escalamento”, ou seja, os dados são centrados na média e dividdidos pelo respectivo desvio padrão (Equação R.3).
Onde sj é o desvio padrão dos dados da coluna “j”.
Os dados autoescalados apresentam média zero e variância igual a “1” e tem como objetivo fazer com que todas as variáveis apresentem o mesmo “peso estatístico”.
Quando as variáveis têm diferentes unidades ou quando a faixa de variação dos dados é grande, recomenda-se o autoescalamento, igualando o impacto de cada uma delas. E assim, minimizando o efeito (a influência) de uma variável dominante nos resultados da análise.
O escalamento pela variância e o autoescalamento tornam os dados "adimensionais", ou seja, com valores invariantes com respeito à unidade utilizada originalmente.
Nota
O pré-processamento mais frequente quando se trabalha com dados espectrais é a centragem dos dados na média.
Nos espectros, o efeito causado pelo escalamento pela variância é que todos os comprimentos de onda terão igual peso, não importando se eles representam um pico, um espalhamento ou simplesmente um ruído de linha de base; consequentemente esse pré-processamento não é aconselhável.
Existem outras variedades de escalamento tais como: escalamento de pareto, escalamento segundo a estabilidade da variável (VAST - variable stability), escalamento por nivelamento e escalamento pela amplitude. (Fonte: Quimiometria - Conceitos, Métodos e Aplicações, 2015)
Vamos usar como exemplo o conjunto de dados com as concentrações, em partes por mil, 0/00, de alguns nutrientes encontrados em água do mar coletadas no litoral norte de São Paulo m 1986. (Fonte: Quimiometria - Conceitos, Métodos e Aplicações, 2015)
A tabela também inclui os valores de temperatura (Temp) em °C e a salinidade em partes por mil, 0/00.
O arquivo de dados está disponível para download no link Characterization_Northern_Sao_Paulo_Coast.csv.
Comandos em Python para exibição dos gráficos com os dados originais e autoescalados.
#!/usr/bin/env python3
# -*- coding: utf8 -*-
#Programa tutorial para PCA
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
pd.set_option('display.width', 250)
np.set_printoptions(linewidth=250)
data = pd.read_csv('Characterization_Northern_Sao_Paulo_Coast.csv', sep=';', decimal=',')
print("Dados:\n", data)
X = data.values[:,:]
print("Dados:\n", X)
list_par = data.columns.tolist()
#ou
#list_par = data.columns.values
x = np.arange(len(list_par))
#x and y must have same first dimension
x = x.reshape(-1,1)
plt.figure(figsize=(12,4))
#Using subplot()
#The argument of the subplot() function is composed of three integers.
#The first number defines how many parts the figure is split into vertically.
#The second number defines how many parts the figure is divided into horizontally.
#The third issue selects which is the current subplot on which you can direct commands.
plt.subplot(1,2,1)
plt.plot(x, X.T)
plt.xticks(x, list_par, rotation='vertical')
plt.ylabel('Resposta')
plt.xlabel('Parâmetro')
####################################################
plt.subplot(1,2,2)
X_scaled = (X - X.mean(axis = 0))/X.std(axis=0, ddof=1)
print("Dados Escalados:\n", X_scaled)
plt.plot(x, X_scaled.T)
plt.xticks(x, list_par, rotation='vertical')
plt.ylabel('Resposta')
plt.xlabel('Parâmetro')
plt.tight_layout()
plt.show()
Dataframe com os dados originais:
Dados:
NO2- NH3 N2 PO4-3 SiO2 O2 Temp Salinidade
0 0.00 1.2 1.48 0.02 1.64 106.00 23.50 35.92
1 0.09 0.5 0.85 0.02 0.43 110.20 22.74 35.25
2 0.01 0.4 0.71 0.07 0.61 111.83 20.98 35.28
3 0.00 0.1 0.29 0.12 0.68 96.48 16.63 35.43
4 0.06 0.1 0.29 0.36 2.44 81.00 15.35 35.49
5 0.31 1.0 2.34 0.47 5.02 76.42 15.57 35.65
6 0.01 0.3 0.54 0.00 1.25 104.50 22.00 35.49
7 0.07 0.5 0.65 0.04 0.43 100.68 21.99 35.32
8 0.06 0.5 0.70 0.08 2.92 96.48 19.74 35.82
9 0.04 0.3 0.57 0.06 1.40 108.60 17.09 35.72
10 0.08 0.6 0.75 0.01 1.01 103.33 23.21 35.63
11 0.00 0.3 0.30 0.01 0.18 101.00 23.16 35.68
12 0.06 0.5 0.68 0.14 3.55 81.92 17.17 35.75
13 0.08 0.6 13.14 0.43 2.68 85.83 14.06 35.38
Matriz com os dados autoescalados:
Dados Escalados: [[-0.79002063 2.31853883 -0.05484874 -0.67698252 -0.0646941 0.73395597 1.17322979 1.71830312] [ 0.35414718 0.02341958 -0.2430845 -0.67698252 -0.92088014 1.09440541 0.94955724 -1.46072711] [-0.66289087 -0.30445459 -0.28491467 -0.37124848 -0.79351362 1.23429412 0.43157871 -1.31838247] [-0.79002063 -1.28807713 -0.41040517 -0.06551444 -0.7439822 -0.08306275 -0.84865233 -0.60665929] [-0.02724209 -1.28807713 -0.41040517 1.40200896 0.50137931 -1.41157639 -1.22536399 -0.32197001] [ 3.15100182 1.66279047 0.20210801 2.07462385 2.32696606 -1.80463792 -1.16061667 0.43720139] [-0.66289087 -0.63232877 -0.33570844 -0.79927613 -0.34065489 0.60522403 0.73177081 -0.32197001] [ 0.09988767 0.02341958 -0.30284188 -0.5546889 -0.92088014 0.27738668 0.72882775 -1.12858962] [-0.02724209 0.02341958 -0.28790254 -0.31010167 0.84102335 -0.08306275 0.06663928 1.243821 ] [-0.2815016 -0.63232877 -0.32674484 -0.43239529 -0.23451613 0.95709133 -0.71327158 0.76933887] [ 0.22701742 0.35129376 -0.27296319 -0.73812933 -0.51047692 0.50481311 1.08788105 0.34230496] [-0.79002063 -0.63232877 -0.4074173 -0.73812933 -1.09777808 0.3048495 1.07316575 0.57954602] [-0.02724209 0.02341958 -0.29387828 0.05677918 1.28680617 -1.3326208 -0.6897271 0.91168351] [ 0.22701742 0.35129376 3.42900672 1.83003661 0.67120133 -0.99705954 -1.60501871 -0.84390035]]
Gráfico com os dados originais à esquerda e após autoescalamento:
Figura R.4. Gráfico dos resultados das análises das amostras de água. Á esquerda os dados originais, e à direita os dados autoescalados. (Fonte: Quimiometria - Conceitos, Métodos e Aplicações, 2015)
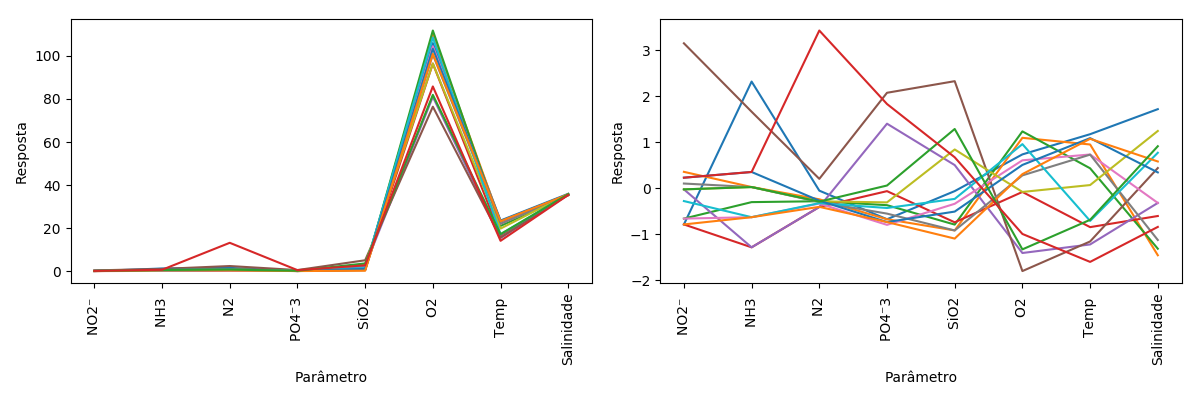
As variáveis O2, Temp e Salinidade possuem valores médios bem mais altos do que as demais.
Mas apesar dos valores absolutos dessas variáveis serem altos o desvio padrão da Salinidade é muito pequeno (0,21).
Por outro lado, as concentrações de N2 são pequenas mas apresentam um desvio padrão relativamente alto (3,34), e da mesma ordem de grandeza do desvio padrão da variável Temp.
Após o autoescalamento, as variáveis com desvio padrão mais alto (N2 3,34 - O2 11,65 e Temp 3,40) são “comprimidas” e aquelas com baixo desvio padrão (NO2- 0,08 - NH3 0,31 - PO4-3 0,16 - Salinidade 0,21) são “expandidas”. E dessa forma, permite que a contribuição desses nutrientes seja tão significativa quanto as demais variáveis.
E portanto todas as variáveis terão igual importância nas análises posteriores independente dos valores absolutos.