Nota
Para uma introdução às técnicas de Análise de Componentes Principais - PCA e Principal Components Regression - PCR, veja a seção Análise de Componentes Principais (PCA) & Modelos de Regressão com Componentes Principais (PCR)
Nessa seção vamos mostrar a aplicação prática da técnica de PCR (Principal Components Regression ou Regressão sobre os Componentes Principais) no desenvolvimento de um modelo de Calibração Multivariada para a determinação da concentração dos componentes de uma mistura de 10 Hidrocarbonetos Poliaromáticos (PAH) utilizando vários comprimentos de onda dos espectros de absorção UV/Vis.
Serão usados como variáveis independentes (X) as leituras de absorbância em diferentes comprimentos de onda dos espectros de UV/Vis das misturas de PAHs.
E como variável dependente (Y) serão usadas as concentrações dos hidrocarbonetos componentes da mistura.
Vamos usar o dataset com 25 espectros de 25 soluções diferentes contendo misturas de 10 hidrocarbonetos poliaromáticos (PAH) com 5 concentrações diferentes.
Onde cada espectro é formado por “27” valores de absorbância entre 220 e 350 nm, com intervalos de 5 nm entre cada leitura.
Os espectros estão disponíveis para download no link tabela_25_espectros_UV_Vis_25_comp_onda_220_350_de_mist_10_PAH.csv.
E a tabela com as 25 amostras com 5 diferentes concentrações dos 10 Hidrocarbonetos Poliaromáticos está disponível para download no link tabela_25_solucoes_10_PAH.csv.
Nos métodos baseados em MLR (Multivariate Linear Regression) é necessário conhecer “todos” os componentes “significativos” da mistura, na região de interesse. Mas os métodos baseados em PCA (Principal Componente Analysis) não exigem detalhes sobre os espectros ou concentrações de todos os compostos em uma mistura, embora seja importante fazer uma estimativa “razoável” de quantos componentes significativos caracterizam uma mistura, mas não necessariamente de suas características químicas. (Fonte: Chemometrics - Data Driven Extraction for Science, 2018)
Vamos primeiro calcular a matriz de scores (T) a partir da tabela de valores de absorbância nos diferentes comprimentos de onda, que são as variáveis “dependentes” (Y).
Mas como vamos usar a técnica de calibração multivariada inversa, essa transformação é feita pelo produto matricial da matriz de absorbâncias com a matriz de “autovetores” (loadings - pesos) que vamos representar por “U”, segundo a equação W.5).
Em seguida vamos calcular as estimativas dos parâmetros (β1, β2 ... β10) do modelo de Regressão por Componentes Principais (PCR), usando a matriz das concentrações (C) como variáveis “independentes” e a matriz de scores (T) como variáveis “dependentes” usando a equação W.6.
Equação W.6. Equação matricial geral para o cálculo da matriz de parâmetros B pela técnica dos “mínimos quadrados” para a “calibração multivariada inversa” usando a matriz de scores (T).
B ≈ (Tt •T)-1 • Tt • C
E finalmente as concentrações estimadas para os componentes da mistura podem ser obtidas pela equação:
Ĉ = T • B
Vamos inicialmente calcular os componentes principais usando os dados “originais” da tabela tabela_25_espectros_UV_Vis_25_comp_onda_220_350_de_mist_10_PAH.csv.
Nota
Os dados poderiam ser pré-processados por escalamento ou centralização, mas vamos usar os dados originais.
Carregando as bibliotecas NumPy, Pandas e Matplotlib.
import numpy as np import pandas as pd import matplotlib.pyplot as plt
Dica
Para melhorar a visualização das matrizes no terminal ajustamos a largura de 200 colunas com o comando:
np.set_printoptions(linewidth=200)
Abrindo o arquivo com os espectros e armazenando como um “dataframe” na variável espectros:
espectros = pd.read_csv ('tabela_25_espectros_UV_Vis_25_comp_onda_220_350_de_mist_10_PAH.csv', sep = ' ', decimal=b'.', skipinitialspace=True)
Abrindo a tabela de concentrações e armazenando na variável conc_solutions:
conc_solutions = pd.read_csv ('tabela_25_solucoes_10_PAH.csv', sep = ',', skiprows = 1, decimal=b',', skipinitialspace=True)
Extraindo os valores de absorbância das colunas 1-27 e armazenando na variável “array” Y:
Y = espectros.values[:,1:28]
print("Y :\n",Y, Y.shape)
Y : [[0.771 0.714 0.658 0.537 0.587 0.671 0.768 0.837 0.673 0.678 0.741 0.755 0.682 0.633 0.706 0.29 0.208 0.161 0.135 0.137 0.162 0.13 0.127 0.165 0.11 0.075 0.053] [0.951 0.826 0.737 0.638 0.738 0.911 1.121 1.162 0.869 0.87 0.965 1.05 0.993 0.934 1.008 0.405 0.254 0.185 0.157 0.159 0.18 0.155 0.15 0.178 0.14 0.105 0.077] [0.912 0.847 0.689 0.514 0.504 0.622 0.805 0.892 0.697 0.728 0.79 0.728 0.692 0.639 0.717 0.292 0.224 0.168 0.134 0.123 0.136 0.115 0.095 0.102 0.089 0.068 0.048] [0.688 0.679 0.662 0.558 0.655 0.738 0.838 0.883 0.67 0.656 0.704 0.668 0.562 0.516 0.573 0.295 0.212 0.172 0.138 0.144 0.179 0.13 0.134 0.191 0.107 0.06 0.046] [0.873 0.801 0.732 0.64 0.75 0.82 0.907 0.955 0.692 0.681 0.775 0.866 0.798 0.749 0.827 0.311 0.213 0.17 0.151 0.162 0.201 0.158 0.17 0.239 0.146 0.094 0.067] [0.953 0.85 0.732 0.612 0.724 0.882 1.013 1.087 0.86 0.859 0.925 0.938 0.869 0.81 0.868 0.39 0.241 0.175 0.138 0.141 0.167 0.129 0.135 0.178 0.115 0.078 0.056] [0.613 0.577 0.547 0.448 0.508 0.463 0.473 0.549 0.51 0.56 0.604 0.493 0.369 0.343 0.363 0.215 0.178 0.165 0.136 0.142 0.183 0.122 0.129 0.193 0.089 0.041 0.03 ] [0.927 0.866 0.799 0.605 0.618 0.671 0.771 0.842 0.646 0.658 0.716 0.829 0.783 0.731 0.794 0.292 0.22 0.155 0.12 0.123 0.144 0.122 0.127 0.164 0.113 0.078 0.056] [0.585 0.577 0.543 0.45 0.515 0.614 0.734 0.815 0.624 0.642 0.724 0.716 0.68 0.649 0.713 0.298 0.213 0.167 0.136 0.126 0.137 0.109 0.104 0.129 0.098 0.074 0.057] [0.835 0.866 0.803 0.55 0.563 0.577 0.642 0.732 0.675 0.713 0.836 0.938 0.911 0.847 0.91 0.358 0.226 0.173 0.153 0.16 0.186 0.156 0.157 0.193 0.134 0.093 0.066] [0.477 0.454 0.45 0.433 0.538 0.593 0.661 0.724 0.554 0.561 0.616 0.469 0.355 0.322 0.345 0.211 0.154 0.139 0.114 0.118 0.154 0.1 0.1 0.154 0.071 0.03 0.016] [0.496 0.45 0.402 0.331 0.389 0.504 0.613 0.599 0.383 0.344 0.353 0.366 0.338 0.309 0.334 0.194 0.123 0.094 0.076 0.07 0.077 0.065 0.056 0.065 0.053 0.036 0.025] [0.594 0.512 0.441 0.392 0.489 0.562 0.637 0.647 0.415 0.387 0.421 0.461 0.396 0.361 0.424 0.161 0.103 0.076 0.062 0.076 0.11 0.082 0.094 0.144 0.078 0.043 0.028] [0.512 0.478 0.409 0.319 0.375 0.395 0.436 0.487 0.439 0.449 0.474 0.438 0.362 0.331 0.36 0.204 0.158 0.122 0.089 0.087 0.107 0.075 0.079 0.114 0.064 0.04 0.028] [0.662 0.583 0.586 0.564 0.687 0.708 0.748 0.757 0.611 0.581 0.641 0.727 0.638 0.596 0.658 0.277 0.171 0.133 0.113 0.128 0.168 0.125 0.143 0.211 0.114 0.067 0.044] [0.768 0.588 0.501 0.386 0.429 0.539 0.671 0.74 0.607 0.627 0.693 0.772 0.751 0.71 0.781 0.287 0.174 0.116 0.093 0.089 0.095 0.087 0.081 0.087 0.081 0.069 0.047] [0.635 0.557 0.487 0.377 0.404 0.488 0.597 0.669 0.603 0.625 0.647 0.576 0.515 0.477 0.524 0.26 0.188 0.139 0.105 0.096 0.104 0.084 0.071 0.077 0.061 0.045 0.031] [0.575 0.489 0.432 0.375 0.408 0.449 0.525 0.569 0.442 0.451 0.501 0.519 0.474 0.458 0.5 0.219 0.151 0.119 0.095 0.092 0.107 0.085 0.081 0.106 0.072 0.047 0.032] [0.768 0.713 0.644 0.528 0.599 0.807 1.009 1.035 0.75 0.722 0.79 0.907 0.921 0.866 0.924 0.375 0.219 0.144 0.118 0.116 0.123 0.12 0.114 0.119 0.115 0.096 0.07 ] [0.811 0.655 0.593 0.496 0.601 0.694 0.81 0.835 0.669 0.671 0.718 0.641 0.566 0.524 0.573 0.29 0.182 0.148 0.123 0.119 0.141 0.103 0.098 0.13 0.08 0.051 0.036] [0.827 0.714 0.66 0.535 0.601 0.66 0.729 0.775 0.619 0.601 0.64 0.667 0.571 0.525 0.602 0.242 0.16 0.12 0.103 0.122 0.164 0.127 0.133 0.182 0.105 0.059 0.037] [0.673 0.492 0.427 0.447 0.584 0.751 0.908 0.931 0.656 0.611 0.623 0.552 0.466 0.418 0.443 0.275 0.199 0.146 0.104 0.094 0.106 0.074 0.07 0.095 0.064 0.042 0.03 ] [0.949 0.852 0.711 0.559 0.586 0.701 0.855 0.907 0.737 0.731 0.801 0.956 0.934 0.881 0.934 0.38 0.239 0.158 0.127 0.125 0.138 0.126 0.124 0.138 0.118 0.093 0.063] [0.939 0.835 0.723 0.622 0.716 0.791 0.908 1.031 0.894 0.948 1.036 1.079 0.948 0.897 0.983 0.386 0.26 0.189 0.15 0.16 0.199 0.155 0.163 0.219 0.145 0.101 0.07 ] [1.055 0.989 0.894 0.681 0.663 0.726 0.837 0.914 0.813 0.84 0.916 0.892 0.837 0.785 0.846 0.359 0.237 0.179 0.151 0.15 0.175 0.145 0.128 0.147 0.116 0.086 0.058]] (25, 27)
Extraindo os valores de concentração das colunas 1-10 e armazenando na variável “array” C:
C = conc_solutions.values[:,1:11]
print("C:\n", C, C.shape)
C: [[0.456 0.12 0.168 0.12 0.336 1.62 0.12 0.6 0.12 0.564] [0.456 0.04 0.28 0.2 0.448 2.7 0.12 0.4 0.16 0.752] [0.152 0.2 0.28 0.16 0.56 1.62 0.08 0.8 0.16 0.118] [0.76 0.2 0.224 0.2 0.336 1.08 0.16 0.8 0.04 0.752] [0.76 0.16 0.28 0.12 0.224 2.16 0.16 0.2 0.16 0.564] [0.608 0.2 0.168 0.08 0.448 2.16 0.04 0.8 0.12 0.94 ] [0.76 0.12 0.112 0.16 0.448 0.54 0.16 0.6 0.2 0.118] [0.456 0.08 0.224 0.16 0.112 2.16 0.12 1. 0.04 0.118] [0.304 0.16 0.224 0.04 0.448 1.62 0.2 0.2 0.04 0.376] [0.608 0.16 0.056 0.16 0.336 2.7 0.04 0.2 0.08 0.118] [0.608 0.04 0.224 0.12 0.56 0.54 0.04 0.4 0.04 0.564] [0.152 0.16 0.168 0.2 0.112 0.54 0.08 0.2 0.12 0.752] [0.608 0.12 0.28 0.04 0.112 1.08 0.04 0.6 0.16 0.376] [0.456 0.2 0.056 0.04 0.224 0.54 0.12 0.8 0.08 0.376] [0.76 0.04 0.056 0.08 0.112 1.62 0.16 0.4 0.08 0.94 ] [0.152 0.04 0.112 0.04 0.336 2.16 0.08 0.4 0.2 0.376] [0.152 0.08 0.056 0.12 0.448 1.08 0.08 1. 0.08 0.564] [0.304 0.04 0.168 0.16 0.224 1.08 0.2 0.4 0.12 0.118] [0.152 0.12 0.224 0.08 0.224 2.7 0.08 0.6 0.04 0.94 ] [0.456 0.16 0.112 0.08 0.56 1.08 0.12 0.2 0.2 0.94 ] [0.608 0.08 0.112 0.2 0.224 1.62 0.04 1. 0.2 0.752] [0.304 0.08 0.28 0.08 0.336 0.54 0.2 1. 0.16 0.94 ] [0.304 0.2 0.112 0.12 0.112 2.7 0.2 0.8 0.2 0.564] [0.76 0.08 0.168 0.04 0.56 2.7 0.16 1. 0.12 0.376] [0.304 0.12 0.056 0.2 0.56 2.16 0.2 0.6 0.08 0.752]] (25, 10)
Y_t = Y.T Y_cov = np.cov(Y_t)
É necessário calcular primeiro a transposta de Y (Y_t) pois o comando np.cov precisa receber um array onde as linhas representam as variáveis (absorbâncias), e as colunas representam as amostras.
Vamos usar o comando np.linalg.eig sobre a matriz de covariância de Y (Y_cov), o qual retorna dois arrays: os autovalores eigen_values e os “respectivos” autovetores eigen_vectors:
eigen_values, eigen_vectors = np.linalg.eig(Y_cov)
Os maiores autovalores indicam os componentes principais (PCs) que mais contribuem para a “variância” dos dados. Mas eles não estão ordenados e, por isso, vamos primeiro ordenar em ordem decrescente para facilitar a seleção dos PCs mais importantes:
idx = eigen_values.argsort()[::-1]
eigen_values = eigen_values[idx]
eigen_vectors = eigen_vectors[:,idx]
print("eigen_values (com 3 casas decimais):\n", np.round(eigen_values, 3), eigen_values.shape)
print("eigen_vectors (com 3 casas decimais):\n", np.round(eigen_vectors, 3), eigen_vectors.shape)
E a saída:
eigen_values (com 3 casas decimais): [ 0.362 0.03 0.018 0.009 0.007 0.002 0.001 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. -0. ] (27,) eigen_vectors (com 3 casas decimais): [[-0.251 -0.107 0.316 -0.149 -0.525 0.607 0.017 0.21 0.211 -0.132 -0.141 -0.129 0.015 -0.077 0.03 -0.009 -0.021 -0.077 -0.043 0.015 0.002 0.019 -0.003 -0.01 -0.009 -0.003 0.002] [-0.238 -0.206 0.404 -0.148 -0.262 -0.291 0.207 0.134 -0.416 0.087 0.398 0.349 0.031 0.08 -0.073 0.028 0.003 0.135 0.076 -0.029 0.013 -0.045 0.015 0.022 0.017 0.012 -0.019] [-0.205 -0.156 0.427 -0.174 0.025 -0.447 -0.179 -0.326 0.016 -0.1 -0.331 -0.478 0.051 0.094 0.06 -0.045 -0.011 -0.093 0.015 -0.069 -0.016 0.044 -0.017 -0.004 0.003 -0.004 0.01 ] [-0.153 0.074 0.288 -0.145 0.191 -0.033 -0.195 -0.182 0.444 0.338 0.049 0.41 -0.32 -0.209 0.007 -0.086 -0.031 -0.017 -0.3 0.123 -0.068 -0.004 0.047 -0.075 -0.036 -0.05 -0.004] [-0.148 0.266 0.234 -0.158 0.406 0.269 -0.207 0.028 0.091 0.028 0.31 -0.088 0.143 0.288 -0.107 0.082 0.145 -0.091 0.504 0.099 0.034 -0.041 0.03 0.086 0.003 0.07 -0.06 ] [-0.187 0.426 -0. -0.204 0.101 0.066 -0.169 -0.037 -0.193 -0.159 0.14 -0.068 0.324 -0.202 0.135 0.102 -0.372 0.312 -0.347 -0.097 0.174 -0.008 -0.151 -0.03 -0.04 -0.007 0.123] [-0.232 0.508 -0.176 -0.197 -0.206 -0.162 0.042 0.066 0.032 -0.219 -0.215 0.185 -0.263 0.42 -0.03 0.02 0.351 0.014 -0.107 -0.161 -0.012 0.023 -0.014 -0.003 0.031 -0.058 0.024] [-0.254 0.433 -0.127 0.004 -0.186 -0.182 0.406 -0.011 0.038 0.291 0.063 -0.27 -0.03 -0.328 -0.001 -0.174 -0.163 -0.191 0.241 0.216 -0.109 -0.004 0.087 0.006 -0.029 0.05 -0.087] [-0.216 0.141 0.045 0.351 -0.07 0.064 -0.301 -0.143 -0.247 0.092 -0.31 0.264 0.197 -0.319 -0.244 0.205 0.099 -0.136 0.149 -0.276 -0.107 -0.032 0.147 0.017 0.175 0.057 -0.125] [-0.226 0.051 0.096 0.529 -0.049 0.071 -0.027 -0.136 -0.051 0.101 -0.067 0.045 0.053 0.178 0.539 -0.074 0.168 0.328 0.065 0.323 -0.052 0.034 -0.105 0.038 -0.043 -0.088 0.115] [-0.26 0.001 0.088 0.536 0.073 -0.005 0.2 -0.151 0.177 -0.317 0.359 -0.105 -0.132 0.122 -0.219 0.088 -0.114 -0.256 -0.242 -0.165 0.174 0.03 0.014 -0.078 -0.083 0.008 -0.008] [-0.329 -0.152 -0.138 0.024 0.214 0.227 0.005 0.087 -0.271 0.328 -0.208 -0.173 -0.381 0.275 -0.229 -0.119 -0.339 0.253 -0.036 -0.118 -0.03 -0.051 0.032 -0.019 0.02 0.049 -0.037] [-0.34 -0.23 -0.285 -0.121 0.048 -0.065 -0.136 0.059 -0.059 -0.105 0.117 -0.176 -0.166 -0.238 -0.107 0.521 0.273 0.043 -0.092 0.424 -0.091 0.006 -0.055 0.013 0.037 -0.038 0.051] [-0.323 -0.234 -0.292 -0.109 0.066 -0.042 -0.113 0.057 0.139 -0.073 0.205 0.031 -0.112 -0.186 0.507 -0.146 0.027 -0.073 0.223 -0.5 0.054 -0.037 0.057 0.082 0.085 0.04 -0.022] [-0.345 -0.252 -0.296 -0.148 0.075 0.035 0.217 -0.246 0.193 0.002 -0.122 0.232 0.56 0.137 -0.236 -0.265 0.061 0.011 -0.043 0.116 0.033 0.017 -0.062 -0.024 -0.062 -0.023 0.023] [-0.108 -0.002 -0.072 0.098 -0.006 -0.137 -0.469 0.376 -0.178 -0.252 0.026 0.09 0. 0.017 -0.043 -0.483 -0.151 -0.304 -0.055 0.322 -0.076 0.089 0.041 -0.054 -0.114 -0.009 -0.04 ] [-0.06 -0.005 0.014 0.123 -0.01 -0.169 -0.089 0.445 0.128 0.443 -0.085 -0.104 0.184 0.026 -0.06 0.127 0.159 -0.142 -0.084 -0.171 0.32 -0.056 -0.1 0.111 -0.321 -0.134 0.353] [-0.036 0.007 0.059 0.127 0.053 -0.168 0.006 0.385 0.263 0.104 0.01 -0.104 0.171 0.043 -0.025 -0.011 0.059 0.172 -0.153 -0.02 -0.009 0.088 -0.298 -0.079 0.507 0.302 -0.432] [-0.033 -0.001 0.058 0.081 0.076 -0.151 0.051 0.268 0.28 -0.19 -0.045 0.007 0.09 -0.047 -0.109 0.079 -0.109 0.392 0.183 -0.097 -0.365 0.187 0.421 -0.291 -0.068 0.021 0.298] [-0.034 -0. 0.082 0.049 0.133 -0.08 0.102 0.176 0.122 -0.211 -0.142 0.027 -0.009 -0.126 -0.066 -0.003 -0.077 0.222 0.08 0.022 0.095 -0.244 0.082 0.346 -0.21 -0.598 -0.38 ] [-0.036 0.012 0.14 0.041 0.221 0.003 0.2 0.113 -0.004 -0.232 -0.154 0.104 -0.101 -0.165 -0.065 -0.106 -0.023 -0.082 0.092 -0.007 -0.253 -0.437 -0.462 0.145 0.205 0.094 0.432] [-0.038 -0.015 0.071 -0.006 0.127 -0.043 0.145 0.054 0.016 -0.189 -0.269 0.194 -0.145 -0.14 0.013 0.051 -0.069 0.086 0.13 0.151 0.448 0.334 0.081 0.359 -0.082 0.49 -0.043] [-0.038 -0.008 0.081 -0.036 0.204 0.045 0.16 0.096 -0.142 -0.08 -0.185 0.043 -0.038 -0.137 0.136 0.007 0.188 -0.033 0.071 0.022 0.248 -0.253 -0.049 -0.71 -0.278 0.085 -0.249] [-0.037 0.021 0.158 -0.055 0.38 0.172 0.293 0.146 -0.27 0.019 -0.021 -0.105 0.095 -0.109 0.138 -0.128 0.352 -0.164 -0.374 -0.106 -0.279 0.298 0.214 0.168 -0.017 -0.056 -0.025] [-0.04 -0.014 0.027 -0.046 0.129 0.008 0.141 0.083 -0.046 0.011 -0.143 0.136 -0.004 0.066 0.134 0.16 -0.245 -0.265 0.125 0.117 0.266 0.305 0.023 -0.218 0.471 -0.426 0.274] [-0.035 -0.025 -0.025 -0.031 0.036 -0.028 0.062 0.059 0.033 0.025 -0.106 0.181 0.056 0.17 0.188 0.375 -0.331 -0.242 0.069 -0.069 -0.409 0.239 -0.358 0.018 -0.39 0.014 -0.255] [-0.026 -0.016 -0.021 -0.02 0.027 -0.049 0.055 0.091 0.042 0.018 -0.099 0.06 0.157 0.249 0.262 0.24 -0.203 -0.214 -0.216 0.159 -0.032 -0.516 0.491 0.113 0.146 0.246 -0.044]] (27, 27)
Para gerar a matriz de “Scores” (T) vamos fazer a multiplicação matricial da matriz Y pela matriz de “Loadings” (U) (variável eigen_vectors) com o comando np.dot:
T = np.dot(Y, eigen_vectors)
print("T (com 4 casas decimais):\n", np.round(T, decimals=4), T.shape)
T (com 4 casas decimais): [[-2.691 0.4319 0.4104 0.04 -0.0257 -0.0226 0.0053 0.0502 0.0152 0.0047 -0.0232 -0.0099 0.0282 -0.0036 -0.0143 -0.0164 -0.0035 -0.0036 0.0021 0.003 0.0013 0.0064 -0.0044 -0.0003 -0.0022 0.0022 0.0001] [-3.5974 0.6201 0.252 0.0286 -0.0351 -0.0072 0.0115 0.0696 0.0219 -0.0143 -0.022 -0.0112 0.0079 0.0085 -0.0116 -0.02 0.0018 -0.0058 0.0036 0.0023 0.0028 0.0053 -0.0046 0.0001 -0.0022 0.0022 0.0001] [-2.7861 0.3855 0.4701 0.0776 -0.2422 -0.0465 0.0465 0.0605 0.0184 0.0017 -0.0047 -0.0017 0.0226 0.0016 -0.0182 -0.0202 0.0082 -0.0076 0.0049 0.0046 0.0015 0.0061 -0.0048 -0. -0.0022 0.0022 0.0001] [-2.5506 0.6524 0.4983 0.0307 0.0146 -0.0741 -0.009 0.0612 -0.0017 0.0053 -0.0248 -0.0161 0.0271 0.0056 -0.0155 -0.0297 0.002 -0.0106 0.0036 0.0038 0.0011 0.0062 -0.0044 0.0002 -0.0022 0.0022 0.0001] [-3.0646 0.5295 0.4486 -0.1148 0.0566 0.0083 0.0259 0.0809 0.0268 -0.0073 -0.0033 -0.009 0.0315 -0.0042 -0.0111 -0.0156 -0.0009 -0.007 0.0066 0.0036 0.0019 0.0066 -0.0045 0.0003 -0.0022 0.0022 0.0001] [-3.3564 0.62 0.3948 0.0705 -0.0884 0.0097 -0.0196 0.0596 -0.0202 -0.0068 0.0117 -0.0179 0.033 -0.0066 -0.0092 -0.0257 0.0015 -0.0049 0.0035 0.0037 0.0022 0.0056 -0.0047 0. -0.0022 0.0022 0.0001] [-1.8777 0.3432 0.5965 0.1403 0.0378 0.0039 0.0019 0.0882 0.0172 -0.0096 -0.0152 -0.0149 0.0154 -0.0019 -0.0011 -0.0202 0.0049 -0.0076 0.0049 0.0045 0.0012 0.0054 -0.0046 -0.0001 -0.0022 0.0022 0.0001] [-2.915 0.2942 0.5023 -0.1151 -0.0934 -0.0224 -0.0137 0.0481 0.0223 0.0339 -0.0066 -0.0357 0.0181 0.0045 -0.0083 -0.0207 0.0043 -0.0063 0.0054 0.0032 0.0022 0.0062 -0.0046 -0. -0.0022 0.0022 0.0001] [-2.5078 0.4131 0.1921 0.1054 0.0324 -0.0785 0.0087 0.0482 0.0333 0.0048 -0.0032 -0.0141 0.0377 0.0017 -0.012 -0.0222 -0.0008 -0.0095 0.0071 0.0027 0.0017 0.0051 -0.0046 -0.0001 -0.0022 0.0022 0.0001] [-3.0246 0.0356 0.3969 0.031 0.0519 -0.0542 -0.0139 0.0647 -0.0082 -0.0225 -0.0077 -0.0246 0.017 0.0007 -0.0205 -0.018 0.0002 -0.0088 0.0053 0.005 0.0024 0.006 -0.0045 -0.0001 -0.0022 0.0022 0.0001] [-1.8859 0.6545 0.4127 0.1525 0.0403 -0.0383 0.0059 0.0274 0.0099 -0.0037 0.0042 -0.0175 0.0019 -0.0057 -0.0154 -0.0205 0.0015 -0.0069 0.0058 0.0022 0.0016 0.0064 -0.0046 0.0001 -0.0022 0.0022 0.0001] [-1.5633 0.4853 0.2914 -0.1017 -0.1242 -0.0555 -0.019 0.0639 0.0009 -0.008 -0.0117 -0.0087 0.017 0.0059 -0.0103 -0.0231 -0.0062 -0.003 0.0067 0.0047 0.0015 0.0059 -0.0048 0. -0.0022 0.0022 0.0001] [-1.7982 0.4899 0.3574 -0.1413 -0.0629 0.0351 0.035 0.0341 -0.0106 -0.0021 0.0021 -0.0096 0.0195 0.0064 -0.0115 -0.0189 0.002 -0.0095 0.0042 0.0039 0.0018 0.0054 -0.0042 -0.0001 -0.0022 0.0022 0.0001] [-1.6068 0.2731 0.3601 0.0916 -0.0444 0.0004 -0.0165 0.0774 -0.0144 0.008 -0.0065 -0.0081 0.0285 0.0064 -0.0117 -0.0144 0.0008 -0.0103 0.0037 0.0015 0.0021 0.0062 -0.0049 0.0001 -0.0022 0.0022 0.0001] [-2.501 0.5041 0.364 -0.0608 0.1376 0.0403 -0.0521 0.0392 0.0215 0.0052 -0.0082 -0.0001 0.0237 0.0034 -0.0175 -0.0195 0.0081 -0.006 0.0047 0.0044 0.0015 0.0057 -0.0048 -0. -0.0022 0.0022 0.0001] [-2.5342 0.2139 0.1233 0.0647 -0.117 0.0711 0.0008 0.0396 0.0235 -0.0105 -0.015 -0.0271 0.0227 -0.0016 -0.013 -0.0214 0.0001 -0.0082 0.0039 0.0038 0.0005 0.0059 -0.0047 0.0002 -0.0022 0.0022 0.0001] [-2.1158 0.3431 0.3143 0.1883 -0.1238 -0.0132 -0.0234 0.0406 0.0048 0.0062 -0.0209 -0.0074 0.0273 -0.0007 -0.0116 -0.0188 0.0054 -0.0037 0.0065 0.0039 0.0028 0.006 -0.0042 0.0002 -0.0022 0.0022 0.0001] [-1.8593 0.274 0.272 0.006 -0.0397 0.0135 -0.0059 0.065 0.0382 0.0005 -0.0035 -0.0039 0.0186 -0.0033 -0.011 -0.0301 -0.0002 -0.0096 0.0033 0.004 0.0031 0.0065 -0.0046 -0.0001 -0.0022 0.0022 0.0001] [-3.1455 0.5267 0.1009 -0.0331 -0.0559 -0.0721 -0.0168 0.0398 -0.0025 -0.0074 -0.015 -0.0082 0.0191 -0.0027 -0.0024 -0.0169 0.0058 -0.0111 0.0049 0.0043 0.0017 0.0066 -0.0046 -0.0001 -0.0022 0.0022 0.0001] [-2.5069 0.5839 0.457 0.0683 -0.1254 0.0341 -0.0268 0.0561 0.022 -0.0339 -0.0065 -0.0199 0.0295 0.0106 -0.0121 -0.0221 0.0041 -0.0079 0.0065 0.0028 0.0018 0.0067 -0.0045 -0.0002 -0.0022 0.0022 0.0001] [-2.4762 0.4578 0.5349 -0.0555 -0.07 0.0431 0.0075 0.033 -0.005 -0.0062 -0.0339 -0.0118 0.0245 -0.0079 -0.0131 -0.0231 0.0003 -0.01 0.007 0.0029 0.0025 0.0057 -0.0048 -0.0001 -0.0022 0.0022 0.0001] [-2.2559 0.8548 0.3078 0.0748 -0.1446 0.0197 -0.0301 0.0837 0.0193 0.0134 -0.0113 -0.0211 0.0186 -0.0031 -0.0171 -0.015 -0.0013 -0.0111 0.0052 0.005 0.0021 0.0057 -0.0045 -0.0001 -0.0022 0.0022 0.0001] [-3.1879 0.2709 0.2843 -0.0354 -0.1102 0.0094 -0.0279 0.0915 -0.0012 0.0075 -0.0078 -0.0023 0.0122 -0.0039 -0.0151 -0.0245 0.0031 -0.0079 0.0068 0.0019 0.0009 0.0059 -0.0043 -0.0001 -0.0022 0.0022 0.0001] [-3.4988 0.4281 0.3442 0.2022 0.0347 0.0614 0.0172 0.0503 -0.0055 0.0198 -0.009 -0.0067 0.018 0.0089 -0.0098 -0.0227 -0.0037 -0.0082 0.0066 0.0048 0.0017 0.0064 -0.0046 -0.0001 -0.0022 0.0022 0.0001] [-3.2745 0.3305 0.6361 0.0499 -0.1506 -0.0276 -0.0443 0.0278 0.0273 -0.0057 -0.0025 0. 0.0178 0.0017 -0.0094 -0.018 -0.0051 -0.0105 0.0044 0.0035 0.0016 0.0057 -0.0045 0.0001 -0.0022 0.0022 0.0001]] (25, 27)
Poderíamos calcular as estimativas dos parâmetros (β1, β2 ... β10) do modelo de Regressão por Componentes Principais (PCR) utilizando “todos” os Componentes Principais com os comandos:
T_t = T.T
T_t_dot_T = np.dot(T_t, T)
inv_T_t_dot_T = np.linalg.inv(T_t_dot_T)
T_t_dot_C = np.dot(T_t, C)
B = np.dot(inv_T_t_dot_T, T_t_dot_C)
print("B: \n", B, B.shape)
B: [[ 0.052 0.016 0.018 0.016 -0.002 -0.851 0.012 0.066 0.023 0.016] [ 0.305 -0.007 0.292 0.002 0.199 -1.093 0.034 0.258 0.022 1.266] [ 0.841 0.092 -0.098 0.219 0.292 -1.216 0.016 0.495 0.035 -0.226] [ -0.1 -0.041 -0.208 -0.135 1.388 -1.332 0.054 0.614 -0.055 -0.359] [ 1.846 -0.148 -0.096 -0.102 -0.273 0.484 0.079 -1.201 -0.22 -0.216] [ 1.319 -0.552 -0.414 -0.519 -0.324 0.748 0.037 1.352 0.893 0.408] [ 1.254 0.143 2.127 -0.103 1.192 1.553 -0.7 -0.278 0.614 -6.187] [ 1.089 1.343 0.54 0.316 -1.214 -2.735 1.311 0.021 1.199 -2.225] [ -3.136 -1.506 0.833 0.514 1.62 -2.87 2.027 -6.967 0.207 -2.912] [ 0.026 -0.53 1.108 -0.596 -3.221 -0.809 1.435 14.184 -1.583 -5.182] [ 1.376 1.29 1.153 -2.204 2.034 -0.196 0.014 -6.946 -0.943 -3.755] [ -2.397 0.698 -1.286 0.444 -0.293 -0.006 1.83 -0.064 0.376 5.164] [ 0.442 3.205 -1.08 -2.531 -0.22 -5.032 0.633 -0.601 -0.374 4.6 ] [ 2.785 1.014 0.285 -0.999 2.443 -4.14 1.34 -7.526 -1.161 -2.061] [ -0.438 -0.551 0.663 -0.208 0.397 6.368 1.136 5.924 0.165 0.628] [ -3.996 -0.605 -0.013 -3.943 0.089 1.594 -1.434 -0.437 -0.315 4.238] [ 1.718 -0.019 0.135 -1.495 -1.861 1.816 -2.774 12.814 -0.044 -4.698] [ -3.805 -1.953 -2.522 3.542 -0.321 -6.718 -6.893 -16.503 1.047 4.611] [ -3.94 3.944 -1.816 -0.584 3.876 27.296 2.154 -4.166 4.557 13.186] [ -2.659 -6.887 3.537 2.504 -0.066 -10.79 -3.229 2.958 -0.497 -28.645] [ -1.775 -24.37 4.712 19.736 0.946 1.614 -11.75 49.956 -14.679 -32.528] [ 11.21 7.282 5.528 -1.92 8.488 44.148 -0.721 1.989 -13.849 21.615] [ 40.613 7.247 29.849 -28.571 -7.423 42.363 46.222 76.724 12.335 -50.954] [ 14.365 -13.672 -1.879 58.509 0.406 13.05 -18.159 1.079 -47.428 74.295] [ -46.822 19.16 -6.447 110.774 85.928 567.561 -85.378 -38.116 -13.446 248.331] [ -154.292 -4.073 40.092 15.198 -15.561 434.769 -40.064 -58.434 4.114 -231.484] [ 479.595 545.977 1506.114 720.673 5535.503 -9383.991 -120.576 3340.794 2565.724 14425.653]] (27, 10)
A matriz B possui 27 linhas e 10 colunas, onde cada coluna corresponde a um dos 10 PAHs e cada linha corresponde a um dos coeficientes do modelo de regressão.
Mas a grande vantagem da técnica de Componentes Principais é a capacidade de “reduzir a dimensionalidade” do modelo de regressão sem perda “significativa” da capacidade de previsão do modelo.
Por isso vamos refazer os cálculos das estimativas dos parâmetros do modelo de Regressão por Componentes Principais (PCR) mas utilizando “apenas” os 10 primeiros Componentes Principais:
T10 = T[:,0:10]
T10_t = T10.T
T10_t_dot_T10 = np.dot(T10_t, T10)
inv_T10_t_dot_T10 = np.linalg.inv(T10_t_dot_T10)
T10_t_dot_C = np.dot(T10_t, C)
B = np.dot(inv_T10_t_dot_T10, T10_t_dot_C)
print("B: \n", B, B.shape)
B: [[-0.043 -0.009 -0.02 -0.008 -0.067 -1.161 -0.009 -0.091 -0.007 -0.093] [ 0.246 -0.022 0.275 -0.017 0.144 -1.366 0.011 0.242 0.015 1.18 ] [ 0.849 0.095 -0.088 0.216 0.282 -1.277 0.007 0.597 0.05 -0.236] [-0.097 -0.04 -0.206 -0.135 1.388 -1.333 0.053 0.634 -0.052 -0.357] [ 1.864 -0.143 -0.091 -0.096 -0.255 0.576 0.088 -1.207 -0.22 -0.188] [ 1.309 -0.555 -0.419 -0.52 -0.329 0.728 0.036 1.324 0.888 0.4 ] [ 1.215 0.132 2.109 -0.111 1.172 1.459 -0.705 -0.378 0.597 -6.225] [ 1.086 1.348 0.587 0.279 -1.34 -3.412 1.23 0.701 1.297 -2.378] [-3.033 -1.478 0.883 0.532 1.666 -2.669 2.034 -6.661 0.258 -2.824] [ 0.026 -0.53 1.106 -0.594 -3.217 -0.785 1.438 14.158 -1.587 -5.177]] (10, 10)
E neste caso obtemos uma matriz B com apenas 10 linhas correspondentes aos 10 parâmetros do modelo de regressão, e 10 colunas correspondentes aos 10 componentes da mistura.
E finalmete vamos o usar o Modelo de Regressão por Componentes Principais (PCR) para estimar as concentrações dos diferentes componentes nas 25 soluções através da equação:
Ĉ = T • B
C_est = np.dot(T10, B)
print("C_est:\n", C_est, C_est.shape)
_est: [[0.505 0.113 0.198 0.13 0.376 1.719 0.127 0.62 0.093 0.425] [0.467 0.122 0.288 0.114 0.454 2.677 0.141 0.376 0.171 0.775] [0.161 0.179 0.296 0.175 0.557 1.643 0.096 0.834 0.164 0.109] [0.682 0.176 0.23 0.164 0.355 1.126 0.12 0.724 0.046 0.734] [0.809 0.128 0.298 0.156 0.22 2.15 0.161 0.311 0.19 0.446] [0.576 0.171 0.159 0.107 0.427 2.236 0.073 0.944 0.147 1.018] [0.78 0.152 0.105 0.153 0.47 0.451 0.164 0.469 0.171 0.163] [0.402 0.111 0.192 0.17 0.097 2.155 0.181 1.017 0.062 0.282] [0.283 0.082 0.235 0.105 0.43 1.679 0.162 0.243 0.019 0.313] [0.577 0.196 0.022 0.157 0.322 2.704 0.075 0.193 0.088 0.259] [0.61 0.077 0.194 0.103 0.549 0.453 0.082 0.473 0.04 0.662] [0.184 0.171 0.183 0.147 0.084 0.562 0.086 0.379 0.1 0.692] [0.556 0.103 0.24 0.091 0.084 1.105 0.005 0.58 0.155 0.48 ] [0.46 0.165 0.067 0.089 0.213 0.63 0.11 0.81 0.112 0.28 ] [0.771 0.019 0.076 0.089 0.115 1.669 0.177 0.394 0.068 0.897] [0.108 0.03 0.099 0.038 0.352 2.203 0.103 0.356 0.186 0.375] [0.178 0.101 0.072 0.085 0.482 1.065 0.109 0.809 0.071 0.482] [0.269 0.065 0.144 0.104 0.222 1.084 0.182 0.268 0.14 0.215] [0.187 0.136 0.217 0.101 0.253 2.616 0.071 0.371 0.036 0.939] [0.405 0.108 0.111 0.144 0.534 1.129 0.11 0.306 0.219 0.974] [0.666 0.109 0.151 0.129 0.285 1.549 0.04 0.729 0.162 0.616] [0.314 0.112 0.259 0.093 0.336 0.499 0.207 0.975 0.163 1.018] [0.326 0.18 0.127 0.116 0.126 2.604 0.163 0.839 0.163 0.507] [0.768 0.077 0.168 0.03 0.524 2.686 0.122 1.141 0.126 0.382] [0.335 0.113 0.055 0.211 0.536 2.138 0.139 0.709 0.09 0.706]] (25, 10)
A “qualidade” ou grau de incerteza do modelo de calibração por PCR pode ser quantificada pelos “resíduos” (erros) que é a diferença entre os valores observados (experimentais) e os valores calculados (estimados) pelo modelo (C - C_est), expresso geralmente pela “Raiz do Erro Médio Quadrático” (Root Mean Square Error - RMSE), calculado pela equação W.7.
Equação W.7. Equação para o cálculo da Raiz do Erro Médio Quadrático (Root Mean Square Error - RMSE) do modelo de Regressão por Componentes Principais (PCR).

Onde ci é a concentração real do componente “c” na solução (amostra) “i”, ĉi é a concentração estimada (calculada) pelo modelo para o componente “c” na solução (amostra) “i”, N é o número de soluções (amostras) (25) e PC é o número de Componentes Principais usados no modelo de regressão (10).
Nota
Se os dados estiverem centralizados, um grau adicional de liberdade será perdido.
A Raiz do Erro Médio Quadrático (RMSE em Inglês) pode ser calculada com os comandos:
SQ_r = np.sum( (C_est - C)**2 , axis = 0)
print("Soma Quadrática dos Resíduos (SQ_r):\n", SQ_r, SQ_r.shape)
n = C.shape[0]
print("Número de amostras: ", n)
pc = 10
print("Número de componentes principais: ", pc)
#Graus de liberdade (gl)
gl = n - pc
print("Graus de liberdade: ", gl)
#Cálculo da Raiz do Erro Médio Quadrático (Root Mean Square Error - RMSE)
rmse = np.sqrt(SQ_r / gl)
print("Raiz do Erro Médio Quadrático:\n", rmse, rmse.shape)
Podemos verificar que o menor erro de previsão foi obtido com o Naftaleno (Nap - 0.02576) e o maior erro de previsão foi obtido com o Fluoreno (Fluore - 0.14724).
Lista de PAHs : ['Py', 'Ace', 'Anth', 'Acy', 'Chry', 'Benz', 'Fluora', 'Fluore', 'Nap', 'Phen'] 10 Lista de RMSE: [0.0469924 0.04396932 0.02635764 0.05043779 0.03067871 0.07198179 0.03798625 0.14724337 0.02576186 0.10817419]
E para exibir em um gráfico com os valores de RMSE para todos os PAHs usamos os comandos:
list_pah = conc_solutions.columns.tolist()
list_pah = list_pah[1:]
print("Lista de PAHs :", list_pah, len(list_pah))
list_rmse = list(rmse)
x = np.arange(len(list_pah))
#Ajuste do tamanho
plt.figure(figsize=(15,6))
plt.subplot(1, 2, 1)
width = 0.3
#Bar graph
plt.bar(x, list_rmse, width, color='blue')
plt.xticks(x, list_pah, rotation='vertical')
plt.ylabel('RMSE')
plt.subplot(1,2,2)
#Scatter graph
plt.plot(x, list_rmse, 's-', color='blue')
plt.xticks(x, list_pah, rotation='vertical')
plt.ylabel('RMSE')
#Set spece between graphs
plt.subplots_adjust(wspace=0.2)
plt.suptitle('Raiz do Erro Médio Quadrático para os PAHs')
plt.show()
Gerando os gráficos de barra e dispersão da figura W.7.
Figura W.7. Gráficos de barras e de dispersão que indicam os valores de Raiz do Erro Médio Quadrático de Previsão para os 10 PAHs, através da técnica de PCR utilizando apenas os 10 primeiros Componentes Principais.
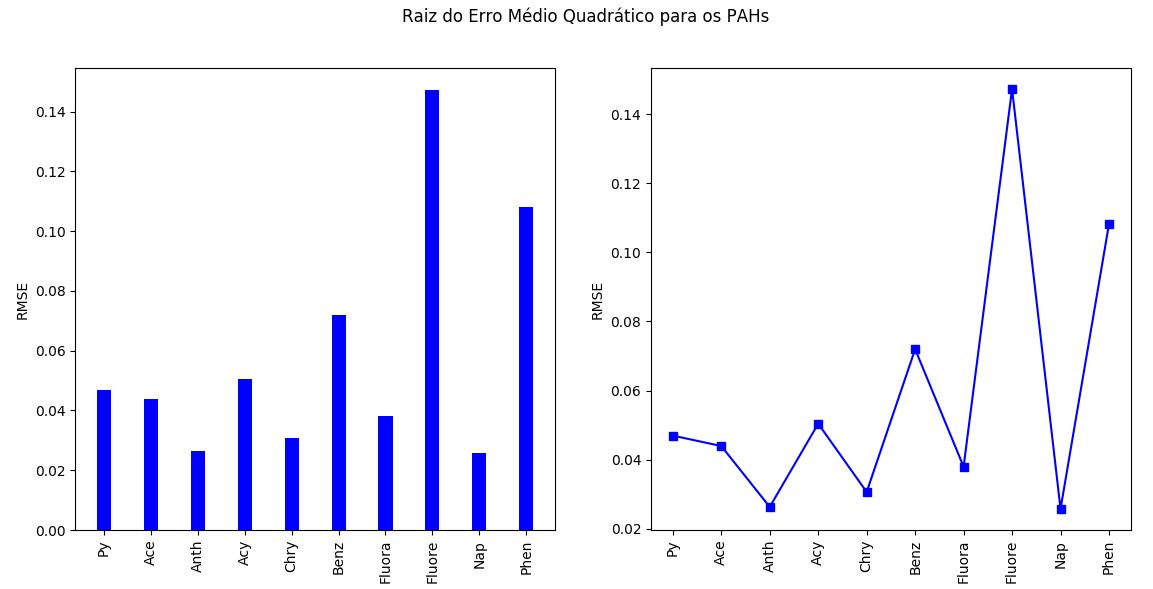
Vamos lembrar que foram usados “27” valores de absorbância, entre 220 e 350 nm, para o cálculo de “27” Componentes Principais (PC).
E o gráfico da figura W.8 mostra, de maneira qualitativa, com aumenta a “capacidade de previsão” da [Pireno] com o modelo por PCR usando 2, 5, 10 e 15 PCs.
Figura W.8. Gráficos das concentrações estimadas de pireno ([Pireno]est) em relação às concentrações reais ([Pireno]real), obtidas pela técnica de PCR utilizando respectivamente 2, 5, 10 e 15 Componentes Principais (PCs) para a calibração.
![Gráficos das concentrações estimadas de pireno ([Pireno]est) em relação às concentrações reais ([Pireno]real), obtidas pela técnica de PCR utilizando respectivamente 2, 5, 10 e 15 Componentes Principais (PCs) para a calibração.](figuras/graf_comp_py_real_py_sim_2_15_pc.png)
Usando novamente o Pireno (Py) como exemplo, adicionamos à figura V.4 o gráfico das concentrações estimadas de pireno ([Pireno]est) em relação às concentrações reais ([Pireno]real), obtidas pela técnica de PCR usando 15 PCs e obtivemos a figura W.9.
Figura W.9. Gráficos das concentrações estimadas de pireno ([Pireno]est) em relação às concentrações reais ([Pireno]real), obtidos respectivamente pelas técnicas de: Calibração Univariada Inversa com 1 comprimento de onda (330nm), Calibração Multivariada Inversa utilizando 4 comprimentos de onda (330, 335, 340 e 345nm) e pela técnica de PCR utilizando 15 Componentes Principais (PCs) para a calibração, calculados a partir de 27 comprimentos de onda entre 220 e 350 nm (com intervalos de 5 nm).
![Gráficos das concentrações estimadas de pireno ([Pireno]est) em relação às concentrações reais ([Pireno]real), obtidos respectivamente pelas técnicas de: Calibração Univariada Inversa com 1 comprimento de onda (330nm), Calibração Multivariada Inversa utilizando 4 comprimentos de onda (330, 335, 340 e 345nm) e pela técnica de PCR utilizando 15 Componentes Principais (PCs) para a calibração, calculados a partir de 27 comprimentos de onda entre 220 e 350 nm (com intervalos de 5 nm).](figuras/graf_comp_cal_univar_multivar_pcr_py.png)
Pode-se verificar qualitativamente, que houve uma melhora na capacidade de previsão do modelo baseado em PCR em relação ao modelo de Calibração Multivariado (Análise de Regressão Multivariada - MLR).
Nota
Os comandos usados nesta seção estão organizados em um único arquivo disponível no link: pca_espectros_00.py.